ESTRUCTURAS
Y
CARACTERÍSTICAS
DEL ADN
Y
CARACTERÍSTICAS
DEL ADN
1- ESTRUCTURA Y FUNCION DEL ADN
- 2. ADN • El ADN es el Ácido Desoxirribonucleico. • Es el tipo de molécula más compleja que se conoce. Contiene la información necesaria para poder controlar el metabolismo un ser vivo. El ADN es el lugar donde reside la información genética de un ser vivo.
- 3. ESTRUCTURA DEL ADN • En 1953, el bioquímico estadounidense James Watson y el biofísico británico Francis Crick publicaron la primera descripción de la estructura del ADN. Su modelo adquirió tal importancia para comprender la síntesis proteica, la replicación del ADN y las mutaciones, que los científicos obtuvieron en 1962 el Premio Nobel de Medicina por su trabajo.
- 4. ESTRUCTURA DEL ADN • Cada molécula de ADN está constituida por dos cadenas o bandas formadas por un elevado número de compuestos químicos llamados nucleótidos. Estas cadenas forman una especie de escalera retorcida que se llama doble hélice.
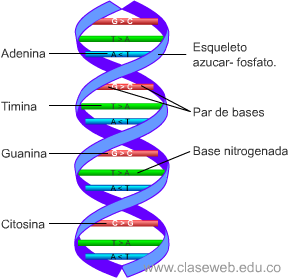
- 5. ESTRUCTURA DEL ADN • Cada nucleótido está formado por tres unidades: una molécula de azúcar llamada desoxirribosa, un grupo fosfato y uno de cuatro posibles compuestos nitrogenados llamados bases: • Adenina (A), Guanina (G), Timina (T) y Citosina (C).
- 6. ESTRUCTURA DEL ADN • La molécula de desoxirribosa ocupa el centro del nucleótido y está flanqueada por un grupo fosfato a un lado y una base al otro. El grupo fosfato está a su vez unido a la desoxirribosa del nucleótido adyacente de la cadena. • Estas subunidades enlazadas desoxirribosa-fosfato forman los lados de la escalera; las bases están enfrentadas por parejas, mirando hacia el interior, y forman los travesaños.
- 7. ESTRUCTURA DEL ADN • Los nucleótidos de cada una de las dos cadenas que forman el ADN establecen una asociación específica con los correspondientes de la otra cadena. • Debido a la afinidad química entre las bases, los nucleótidos que contienen adenina se acoplan siempre con los que contienen timina, y los que contienen citosina con los que contienen guanina. Las bases complementarias se unen entre sí por enlaces químicos débiles llamados enlaces de hidrógeno.
- 8. FUNCIONES DEL ADN: • Las funciones biológicas del ADN incluyen: • Almacenamiento de información (genes y genoma) • Codificación de proteínas (transcripción y traducción) • Autoduplicación (replicación del ADN) Para asegurar así la transmisión de la información a las células hijas durante la división celular.
- 9. FUNCIONES DEL ADN ALMACENAMIENTO DE INFORMACION (GENES Y GENOMAS) • El ADN es la molécula que codifica las instrucciones para crear un ser vivo casi igual a aquél que le da origen. Todas las células que forman a un organismo tienen la misma información genética. Esta cualidad, la de hacer copias exactas de sí mismo, es una característica esencial del material genético y está relacionada con la replicación del ADN.
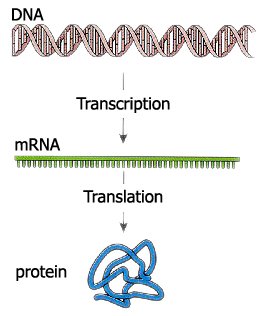
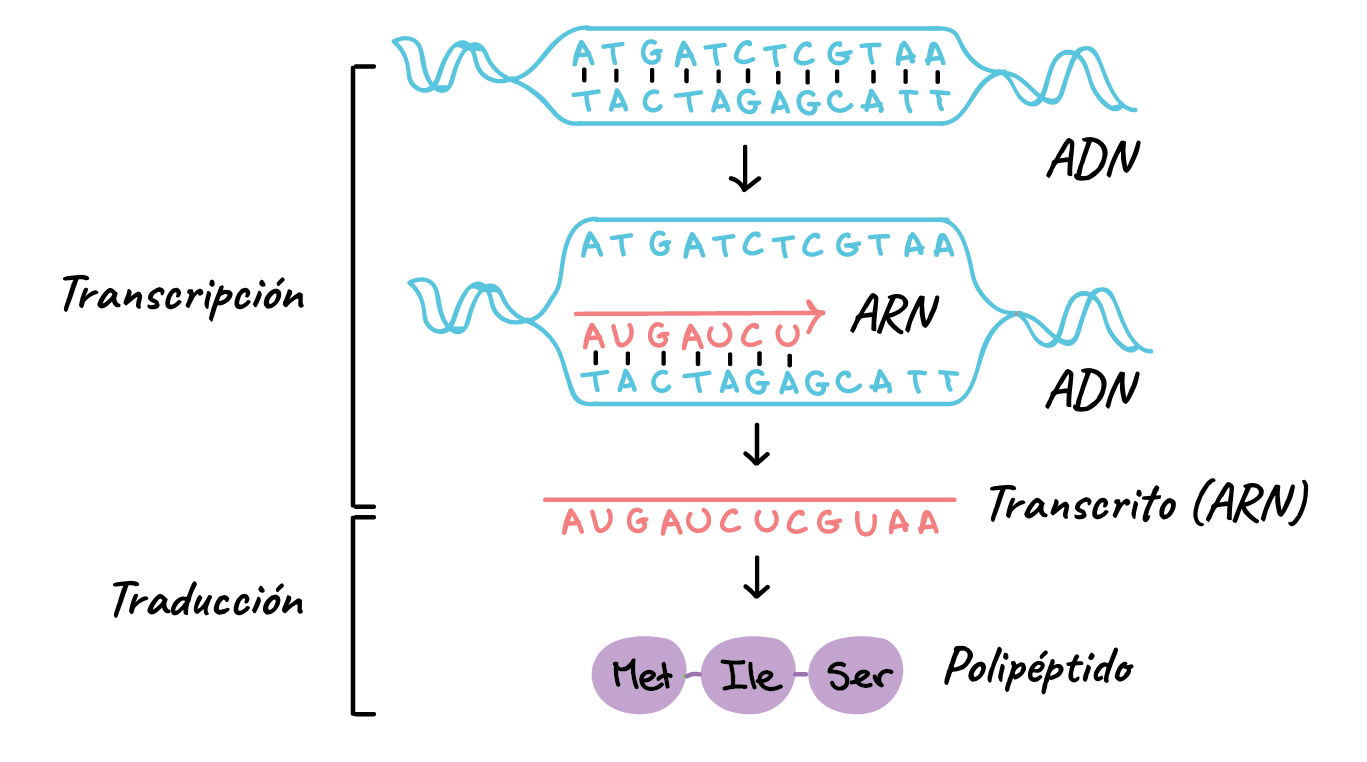
- 10. FUNCIONES DEL ADN CODIFICACION DE PROTEINAS TRANSCRIPCION: • Es el proceso por el que se transmite la información contenida en el ADN al ARN. Este proceso se lleva a cabo por la ARN polimerasa que utiliza como molde una de las dos hebras del ADN, la denominada hebra codificante. Durante el proceso de transcripción se reconoce un sitio específico de la molécula de ADN en el que se van a unir las enzimas. TRADUCCION: • Es el proceso por el que la información genética contenida en el ADN y transcrita en un ARN mensajero va a ser utilizada para sintetizar una proteína. El proceso se lleva a cabo en los ribosomas.
- 11. La transcripción del ADN: • Es el proceso por el que se transmite la información contenida en el ADN al ARN. Este proceso se lleva a cabo por la ARN polimerasa que utiliza como molde una de las dos hebras del ADN, la denominada hebra codificante. Durante el proceso de transcripción se reconoce un sitio específico de la molécula de ADN en el que se van a unir las enzimas.
- 12. La traducción del ADN Es el proceso por el que la información genética contenida en el ADN y transcrita en un ARN mensajero va a ser utilizada para sintetizar una proteína. El proceso se lleva a cabo en los ribosomas.
- 13. FUNCIONES DEL ADN REPLICACIÓN DEL ADN • Durante este proceso, las dos cadenas originales se separan en los puentes hidrógeno, entonces cada una, por separado sirve de molde a partir del cual dos nuevas hebras complementarias se forman con nucleótidos disponibles en la célula. A este modo de duplicación se lo llama modelo semiconservativo.

► "Flujo de la información genética" ◄
I- Introducción:
La transmisión de información genética tiene como principio rector las reglas de apareamiento de bases (Watson-Crick), y es lo que posibilita una transmisión de "ida y vuelta" de la información entre ADN a ARN. De ARN a proteínas, no obstante, la vía de transmisión es unívoca. La replicación es el modo de perpetuar la información genética, y asegurar una copia fiel de la información en cada una de las células producidas por división. En lo referente a la transmisión de la información dentro de la célula, los pasos fundamentales son dos.
El primer paso, la transcripción, consiste en la copia exacta de una de las hebras de ADN a ARN; la secuencia de ARN será exactamente igual a la del ADN copiado, excepto por la presencia de uracilo (U) en vez de timina (T).
El segundo paso, la traducción, implica la síntesis de proteínas haciendo uso del código genético, que identifica aminoácidos específicos a partir de un conjunto de tres bases.
Los tres procesos mencionados son procesos de polimerización, que pueden dividirse en tres etapas: Iniciación, elongación y terminación, definidas en cada caso por eventos concretos.
Entre la transcripción y la traducción, hay en ciertos casos un procesamiento de los transcriptos a fin de obtener ARN mensajero (ARNm) maduro. Los productos de la traducción también son procesados. En cada caso hay en juego elementos de señalización en la molécula que porta la información (ADN, ARN o proteína) para dar lugar a un copiado o procesamiento correcto.
II-
REPLICACIÓN:
La replicación es el modo de perpetuar la información genética, y asegurar una copia fiel de la información en cada una de las células producidas por división. Se divide en tres etapas: Iniciación, elongación y terminación.
Iniciación:
La iniciación de la replicación se da en un origen concreto, y todo ADN replicado a partir de un origen dado se define como replicón. En bacterias, el replicón corresponde al genoma bacteriano, mientras que un cromosoma eucariota contiene varios replicones. A nivel del origen se produce la separación de las dos hebras, mediante enzimas conocidas como helicasas. La reasociación de las hebras separadas es evitada mediante la unión de proteínas SSB (single-stranded DNA binding - de unión a ADN de hebra simple). Para dar inicio efectivo a la replicación, es preciso un grupo 3' OH libre a partir del cual agregar nucleótidos. No existen tales de grupos al momento de la separación de las hebras. En cambio es utilizado el extremo 3' OH libre de un cebador de ARN, que es sintetizado por enzimas conocidas como primasas a fin de dar lugar a un inicio de replicación.
Elongación: Durante la elongación, la polimerización del ADN se produce en dirección 5'-3', en la hebra sintetizada, a través de la unión del oxígeno 3' de la hebra en síntesis al fósforo a del dNTP entrante. Las DNA polimerasas (enzimas capaces de polimerizar ADN) sólo pueden elongar hebras existentes; son incapaces de iniciar una nueva hebra (Por contraste, las RNA polimerasas son capaces de iniciar cadenas polinucelotídicas). El tramo necesario para que las DNA polimerasas comiencen a actuar es proporcionado por el cebador arriba mencionado. La replicación se da en horquillas, cuyas ramas difieren en la dirección de síntesis de ADN. La horquilla de replicación, conteniendo ambas hebras replicadas, se mueve en una dirección única, pero la replicación sólo es capaz de proceder en la dirección 5' - 3'. Esta paradoja es resuelta por el uso de fragmentos de Okazaki: Productos cortos de replicación que se producen en la hebra lagging (o "retrasada"). La otra hebra (leading, o "líder") es replicada en forma continua. De modo que la dirección neta de replicación en la horquilla se mantiene, si bien las hebras obedecen a direcciones distintas.
Terminación:
La terminación obedece, en bacterias, a la presencia de secuencias específicas que dan lugar a la inhibición de la actividad de las helicasas. Estas secuencias se encuentran diametralmente opuestas a los orígenes de replicación en el genoma circular de E. coli. El proceso de terminación en eucariotas es poco conocido. El producto final no contiene ARN, el cual es eliminado a través de la acción de exonucleasa 5'-3'de la DNA polimerasa I, que a su vez rellena con ADN los espacios dejados por el cebador. Por último, la enzima DNA ligasa establece los enlaces azúcar-fosfato entre los tramos replicados y los dejados por la sustitución de los cebadores.
Iniciación:
La iniciación de la replicación se da en un origen concreto, y todo ADN replicado a partir de un origen dado se define como replicón. En bacterias, el replicón corresponde al genoma bacteriano, mientras que un cromosoma eucariota contiene varios replicones. A nivel del origen se produce la separación de las dos hebras, mediante enzimas conocidas como helicasas. La reasociación de las hebras separadas es evitada mediante la unión de proteínas SSB (single-stranded DNA binding - de unión a ADN de hebra simple). Para dar inicio efectivo a la replicación, es preciso un grupo 3' OH libre a partir del cual agregar nucleótidos. No existen tales de grupos al momento de la separación de las hebras. En cambio es utilizado el extremo 3' OH libre de un cebador de ARN, que es sintetizado por enzimas conocidas como primasas a fin de dar lugar a un inicio de replicación.
Elongación: Durante la elongación, la polimerización del ADN se produce en dirección 5'-3', en la hebra sintetizada, a través de la unión del oxígeno 3' de la hebra en síntesis al fósforo a del dNTP entrante. Las DNA polimerasas (enzimas capaces de polimerizar ADN) sólo pueden elongar hebras existentes; son incapaces de iniciar una nueva hebra (Por contraste, las RNA polimerasas son capaces de iniciar cadenas polinucelotídicas). El tramo necesario para que las DNA polimerasas comiencen a actuar es proporcionado por el cebador arriba mencionado. La replicación se da en horquillas, cuyas ramas difieren en la dirección de síntesis de ADN. La horquilla de replicación, conteniendo ambas hebras replicadas, se mueve en una dirección única, pero la replicación sólo es capaz de proceder en la dirección 5' - 3'. Esta paradoja es resuelta por el uso de fragmentos de Okazaki: Productos cortos de replicación que se producen en la hebra lagging (o "retrasada"). La otra hebra (leading, o "líder") es replicada en forma continua. De modo que la dirección neta de replicación en la horquilla se mantiene, si bien las hebras obedecen a direcciones distintas.
Terminación:
La terminación obedece, en bacterias, a la presencia de secuencias específicas que dan lugar a la inhibición de la actividad de las helicasas. Estas secuencias se encuentran diametralmente opuestas a los orígenes de replicación en el genoma circular de E. coli. El proceso de terminación en eucariotas es poco conocido. El producto final no contiene ARN, el cual es eliminado a través de la acción de exonucleasa 5'-3'de la DNA polimerasa I, que a su vez rellena con ADN los espacios dejados por el cebador. Por último, la enzima DNA ligasa establece los enlaces azúcar-fosfato entre los tramos replicados y los dejados por la sustitución de los cebadores.
III- TRANSCRIPCIÓN:


Iniciación: La RNA polimerasa reconoce el promotor y se separan las hebras del ADN a transcribir.
Elongación: La RNA polimerasa polimeriza el transcripto de acuerdo a la secuencia del molde de ADN.reconoce el promotor y se separan las hebras del ADN a transcribir.
Terminación: La RNA polimerasa y el transcripto son liberados tras el reconocimiento de señales específicas.
Se puede definir la transcripción como un proceso de polimerización en la cual nucleótidos individuales se unen formando una cadena de ARN, usando como molde una hebra de ADN. Este proceso, al igual de la replicación y la traducción, tiene tres etapas: Iniciación, elongación y terminación.
La iniciación de la transcripción se da en el momento en que una RNA polimerasa se une al molde de ADN y reconoce la primera base a ser copiada. Esta unión se da en una región específica del ADN conocida como promotor. Esta región contiene elementos de secuencia que se unen a varios factores de transcripción en conjunto a la RNA polimerasa, y que indican la primera base a ser copiada como transcripto. El promotor también incluye secuencias involucradas en la regulación de la transcripción.
En bacterias, el reconocimiento del promotor lo proporciona el factor sigma; en eucariotas, una serie de factores de transcripción llamados factores de iniciación. Entanto el factor sigma forma parte de la holoenzima de la RNA polimerasa en bacterias, los factores de iniciación se unen primero al promotor, formando un complejo que contacta uno o varios sitios de la RNA polimerasa. Una vez unida la RNA polimerasa al ADN, se produce un desenrollamiento del ADN en el promotor.
De las hebras de ADN expuestas al momento del inicio de la transcripción, sólo una contiene la secuencia correcta, actuando de molde. La hebra complementaria no contiene infromación, pero es llamada hebra codificante, en la medida en que su secuencia es idéntica a la de la hebra de ARN sintetizada durante la transcripción. No siempre es la misma hebra la usada de molde: Genes diferentes pueden usar hebras diferentes.
La elongación consiste en el agregado de nucleótidos en forma secuencial tras el agregado de la primera base, hasta que la polimerasa alcance el fin del molde a transcribir. en cada paso de adición, la enzima 1) captura un nucleósido trifosfato y lo aparea al molde; 2) libera dos grupos fosfato del nucleótido; 3) se mueva a la siguiente base a ser copiada. El proceso se repite con el agregado de nucleótidos de a uno, en una dirección que, al igual que la replicación, tiene una dirección 5' - 3', en la que el extremo final de la hebra en construcción está señalada por un OH en 3'.
La terminación, evento final de la transcripción, tiene características distintas en procariotas y eucariotas. En procariotas, consiste en la detención de la elongación y la liberación por parte de la RNA polimerasa del ARN sintetizado. En eucariotas, se observa corte del transcripto, que es inmediatamente poliadenilado en el extremo 3' tratándose de genes transcriptos por la RNA polimerasa II. En ambos casos, hay secuencias que sirven de señal para la terminación, que en algunos casos requieren de factores de terminación específicos.
IV- PROCESAMIENTO DE ARN:
La iniciación de la transcripción se da en el momento en que una RNA polimerasa se une al molde de ADN y reconoce la primera base a ser copiada. Esta unión se da en una región específica del ADN conocida como promotor. Esta región contiene elementos de secuencia que se unen a varios factores de transcripción en conjunto a la RNA polimerasa, y que indican la primera base a ser copiada como transcripto. El promotor también incluye secuencias involucradas en la regulación de la transcripción.
En bacterias, el reconocimiento del promotor lo proporciona el factor sigma; en eucariotas, una serie de factores de transcripción llamados factores de iniciación. Entanto el factor sigma forma parte de la holoenzima de la RNA polimerasa en bacterias, los factores de iniciación se unen primero al promotor, formando un complejo que contacta uno o varios sitios de la RNA polimerasa. Una vez unida la RNA polimerasa al ADN, se produce un desenrollamiento del ADN en el promotor.
De las hebras de ADN expuestas al momento del inicio de la transcripción, sólo una contiene la secuencia correcta, actuando de molde. La hebra complementaria no contiene infromación, pero es llamada hebra codificante, en la medida en que su secuencia es idéntica a la de la hebra de ARN sintetizada durante la transcripción. No siempre es la misma hebra la usada de molde: Genes diferentes pueden usar hebras diferentes.
La elongación consiste en el agregado de nucleótidos en forma secuencial tras el agregado de la primera base, hasta que la polimerasa alcance el fin del molde a transcribir. en cada paso de adición, la enzima 1) captura un nucleósido trifosfato y lo aparea al molde; 2) libera dos grupos fosfato del nucleótido; 3) se mueva a la siguiente base a ser copiada. El proceso se repite con el agregado de nucleótidos de a uno, en una dirección que, al igual que la replicación, tiene una dirección 5' - 3', en la que el extremo final de la hebra en construcción está señalada por un OH en 3'.
La terminación, evento final de la transcripción, tiene características distintas en procariotas y eucariotas. En procariotas, consiste en la detención de la elongación y la liberación por parte de la RNA polimerasa del ARN sintetizado. En eucariotas, se observa corte del transcripto, que es inmediatamente poliadenilado en el extremo 3' tratándose de genes transcriptos por la RNA polimerasa II. En ambos casos, hay secuencias que sirven de señal para la terminación, que en algunos casos requieren de factores de terminación específicos.
IV- PROCESAMIENTO DE ARN:
Muchos transcriptos de ARN son modificados antes de que funcionen correctamente en la célula. Se conocen distintos tipos de modificación:
- Splicing;
- Escisión nucleolítica;
- Agregado de cap;
- Poliadenilado;
- Modificación de bases y azúcares;
- "Edición"
Splicing
Por splicing se entiende una serie de reacciones que dan lugar a la eliminación de intrones del transcripto primario. Estas reacciones consisten en la utilización de un enlace fosfodiéster para la generación de otro.
En el splicing característico de los intrones del Grupo II, el grupo hidroxilo 2' de una adenina en el intrón ataca la unión intrón-exón ubicada a 5'. El hidroxilo 3' liberado del exón ataca luego la unión exón-intrón ubicada a 3', completándose la reacción y liberando el intrón en forma de lazo (lariat).
En el splicing característico de los intrones del Grupo I, el grupo hidroxilo de un nucleósido de guanina ataca la unión intrón-exón ubicada a 5'. El hidroxilo 3' liberado del exón ataca luego la unión exón-intrón ubicada a 3', completándose la reacción. El intrón liberado es capaz de comletar subsiguintes reacciones de transesterificación.
Ambos mecanismos tienen, entonces, dos pasos esenciales. El primero es la ruptura de la unión intrón-exón a 5', seguido por la ruptura respectiva en la unión ubicada a 3'.
El splicing de intrones nucleares no precisa de nucleósidos libres, ni de una estructura secundaria conservada del ARN considerado. No obstante, requiere de un complejo consistente en 44 o más porteínas y una serie de ARN nucleares pequeños (snRNAs - small nuclear RNAs). Éstos juegan el rol de las estructuras secundarias de los intrones de los grupos I y II.
El splicing de intrones nucleares requiere secuencias internas específicas, enunciadas por la "regla GT-AG", que describen los extremos del intrón. En forma adicional, se halla un "sitio de ramificación", con la secuencia UACUAAC en levaduras (menos conservada en mamíferos).
Al igual que las reacciones autónomas descriptas para los intrones de los grupo I y II, el extremo 5' del intrón es el primero en ser liberado, produciéndose una reacción entre el exón a 5' y la unión intrón-exón ubicado a 3'. Al igual que el tipo II, se produce un lazo por la reacción entre el extremo 3' del intrón y el sitio de ramificación.
Escisión nucleolítica
Los genes de algunos ARN de transferencia contienen 14 a 20 nucleótidos en el brazo del anticodón que no están presentes en el ARNt maduro. Los transcriptos conteniendo intrones son sustrato de una endonucleasa que escinde el ARN en cada extremo del intrón. La estructura secundaria de los precursores de ARNt mantiene próximos los exones a 5' y 3' tras el corte.
Los extremos sueltos del ARNt cortado son ligados en una reacción que requiere ATP. Posteriormente, se da una modificación de algunas bases a fin de completar la maduración.
A diferencia de los intrones quitados por splicing, el procesamiento de los precursores de ARNt no involura transesterificación, y es precisa una fuente externa de energía.
El corte de un transcripto por una endonucleasa es también característico de los precursores de ARN ribosomal: En procariotas, los extremos 5' de los ARNr 16S y 23S son generados por la RNAsa C, una endonucleasa. De los ITS (Internal Transcribed Spacers - espaciadores internos transcriptos) se generan precursores de ARNt. Un proceso semejante se da en Eucariotas. En muchos casos, el correcto procesamiento depende de estructuras secundarias de los ARN involucrados.
Varios ARN maduros pueden obtenerse del mismo transcripto. Un buen ejemplo de ello es el genoma mitocondrial, que contiene un número reducido de promotores; de sus transcriptos se generan ARN estructurales, ribosomales y de transferencia.
Modificaciones de los extremos del ARN mensajero
Los extremos 5' de transcriptos de RNA polimerasa II tienen estructuras especiales llamadas caps. En la mayor parte de los eucariotas, se trata de una 7 - metilguanosina trifosfato, que es agregada poco después de la iniciación de la transcripción. Esto aumenta enormemente la traducibilidad de estos ARNm, dado que el cap estimula la unión de ciertos factores de traducción.
La poliadenilación es el agregado de una cadena de ácido poliadenílico (poliA) de 50 a 200 nucleótidos en el extremo 3' del precursor de ARN mensajero. Esto requiere de un corte previo. La señal consenso aislada es AAUAAA, a unos 10-30 nucleótidos 5' del sitio poliA. Igualmente, se ha observado un elemento rigo en GU o U a 3'.
Modificación de bases y azúcares
Algunas bases y azúcares en los ARN llevan modificaciones, como ser metilación. La metilación afecta residuos de adenina en ARNr, la guanina del cap, y el 2' OH de ribosas, presentes cerca del cap en transcriptos de RNA polimerasa y algunas posiciones del ARNr.
Los ARNt en particular contienen una amplia variedad de bases modificadas, creadas por distintas enzimas.
Las modificaciones proveen de una mayor riqueza de señales, más allá de los cuatro nucleótidos básicos. En el caso de ARN mensajeros, no afectan la capacidad codificante de éstos, si bien existen cambios que afectan los patrones de lectura.
"Edición" del ARN
En ciertos casos, se ha observado diferencias notables entre el transcripto primario y el ARN mensajero a traducir. Esas diferencias son debidas a procesos de "edición". Esta edición puede realizarse de dos modos:
Por splicing se entiende una serie de reacciones que dan lugar a la eliminación de intrones del transcripto primario. Estas reacciones consisten en la utilización de un enlace fosfodiéster para la generación de otro.
En el splicing característico de los intrones del Grupo II, el grupo hidroxilo 2' de una adenina en el intrón ataca la unión intrón-exón ubicada a 5'. El hidroxilo 3' liberado del exón ataca luego la unión exón-intrón ubicada a 3', completándose la reacción y liberando el intrón en forma de lazo (lariat).
En el splicing característico de los intrones del Grupo I, el grupo hidroxilo de un nucleósido de guanina ataca la unión intrón-exón ubicada a 5'. El hidroxilo 3' liberado del exón ataca luego la unión exón-intrón ubicada a 3', completándose la reacción. El intrón liberado es capaz de comletar subsiguintes reacciones de transesterificación.
Ambos mecanismos tienen, entonces, dos pasos esenciales. El primero es la ruptura de la unión intrón-exón a 5', seguido por la ruptura respectiva en la unión ubicada a 3'.
El splicing de intrones nucleares no precisa de nucleósidos libres, ni de una estructura secundaria conservada del ARN considerado. No obstante, requiere de un complejo consistente en 44 o más porteínas y una serie de ARN nucleares pequeños (snRNAs - small nuclear RNAs). Éstos juegan el rol de las estructuras secundarias de los intrones de los grupos I y II.
El splicing de intrones nucleares requiere secuencias internas específicas, enunciadas por la "regla GT-AG", que describen los extremos del intrón. En forma adicional, se halla un "sitio de ramificación", con la secuencia UACUAAC en levaduras (menos conservada en mamíferos).
Al igual que las reacciones autónomas descriptas para los intrones de los grupo I y II, el extremo 5' del intrón es el primero en ser liberado, produciéndose una reacción entre el exón a 5' y la unión intrón-exón ubicado a 3'. Al igual que el tipo II, se produce un lazo por la reacción entre el extremo 3' del intrón y el sitio de ramificación.
Escisión nucleolítica
Los genes de algunos ARN de transferencia contienen 14 a 20 nucleótidos en el brazo del anticodón que no están presentes en el ARNt maduro. Los transcriptos conteniendo intrones son sustrato de una endonucleasa que escinde el ARN en cada extremo del intrón. La estructura secundaria de los precursores de ARNt mantiene próximos los exones a 5' y 3' tras el corte.
Los extremos sueltos del ARNt cortado son ligados en una reacción que requiere ATP. Posteriormente, se da una modificación de algunas bases a fin de completar la maduración.
A diferencia de los intrones quitados por splicing, el procesamiento de los precursores de ARNt no involura transesterificación, y es precisa una fuente externa de energía.
El corte de un transcripto por una endonucleasa es también característico de los precursores de ARN ribosomal: En procariotas, los extremos 5' de los ARNr 16S y 23S son generados por la RNAsa C, una endonucleasa. De los ITS (Internal Transcribed Spacers - espaciadores internos transcriptos) se generan precursores de ARNt. Un proceso semejante se da en Eucariotas. En muchos casos, el correcto procesamiento depende de estructuras secundarias de los ARN involucrados.
Varios ARN maduros pueden obtenerse del mismo transcripto. Un buen ejemplo de ello es el genoma mitocondrial, que contiene un número reducido de promotores; de sus transcriptos se generan ARN estructurales, ribosomales y de transferencia.
Modificaciones de los extremos del ARN mensajero
Los extremos 5' de transcriptos de RNA polimerasa II tienen estructuras especiales llamadas caps. En la mayor parte de los eucariotas, se trata de una 7 - metilguanosina trifosfato, que es agregada poco después de la iniciación de la transcripción. Esto aumenta enormemente la traducibilidad de estos ARNm, dado que el cap estimula la unión de ciertos factores de traducción.
La poliadenilación es el agregado de una cadena de ácido poliadenílico (poliA) de 50 a 200 nucleótidos en el extremo 3' del precursor de ARN mensajero. Esto requiere de un corte previo. La señal consenso aislada es AAUAAA, a unos 10-30 nucleótidos 5' del sitio poliA. Igualmente, se ha observado un elemento rigo en GU o U a 3'.
Modificación de bases y azúcares
Algunas bases y azúcares en los ARN llevan modificaciones, como ser metilación. La metilación afecta residuos de adenina en ARNr, la guanina del cap, y el 2' OH de ribosas, presentes cerca del cap en transcriptos de RNA polimerasa y algunas posiciones del ARNr.
Los ARNt en particular contienen una amplia variedad de bases modificadas, creadas por distintas enzimas.
Las modificaciones proveen de una mayor riqueza de señales, más allá de los cuatro nucleótidos básicos. En el caso de ARN mensajeros, no afectan la capacidad codificante de éstos, si bien existen cambios que afectan los patrones de lectura.
"Edición" del ARN
En ciertos casos, se ha observado diferencias notables entre el transcripto primario y el ARN mensajero a traducir. Esas diferencias son debidas a procesos de "edición". Esta edición puede realizarse de dos modos:
- Una o varias bases pueden haber sido cambiadas por otras, lo cual altera el mensaje por cambios en los codones individuales.
- Puede haber habido inserción o eliminación de uno o varios nucelótidos, lo cual conduce a cambios en el marco de lectura del mensajero.
V- TRADUCCIÓN:
La traducción es el proceso de convertir la secuencia de bases del ARN mensajero en la secuencia de aminoácidos del polipéptido.
Hay una serie de componentes necesarios para que se efectúe la traducción. El molde lo constituye el ARN mensajero (ARNm), producto de la transcripción y el splicing, y que contiene le secuencia de inserción de aminoácidos. La secuencia es codificada en forma de de tripletes sin puntuación. En el ARN mensajero se encuentran también las señales de inicio (a 5') y terminación (a 3') de la cadena polipeptídica.
Las moléculas responsables de la traducción son los ARN de transferencia (ARNt), de los cuales hay al menos uno por cada uno de los 20 aminoácidos comunes. En un extremo, los ARNt presentan tres nucleótidos, el anticodón, que se aparea en forma específica con un grupo específico de codones. En el extremo opuesto al anticodón hay un sitio para la unión covalente de un aminoácido.
La producción de aminoacil-tRNA (ARNt con su aminoácido correspondiente) corre por cuenta de aminoacil tRNA sintetasas. Hay al menos 20 tipos, y cada tipo activa y une con su aminoácido específico a un tipo - o serie de tipos - de ARNt.
Los ribosomas son agregados de ARN y proteínas. Se pueden separar en dos subunidades, llamadas subunidad mayor y menor. Los ribosomas contienen sitios de unión al ARNm, al ARNt y a una serie de factores proteicos. En los ribosomas se cataliza la formación del enlace peptídico entre el polipéptido en formación y el aminoácido entrante.
Una serie de factores proteicos, usualmente unidos a los ribosomas, son reuqeridos para la correcta iniciación de la síntesis, para la unión de aminoacil tRNAs entrantes y para el movimiento del ribosoma respecto al ARNm tras cada nuevo agregado de aminoácido (translocación).
Hay una serie de componentes necesarios para que se efectúe la traducción. El molde lo constituye el ARN mensajero (ARNm), producto de la transcripción y el splicing, y que contiene le secuencia de inserción de aminoácidos. La secuencia es codificada en forma de de tripletes sin puntuación. En el ARN mensajero se encuentran también las señales de inicio (a 5') y terminación (a 3') de la cadena polipeptídica.
Las moléculas responsables de la traducción son los ARN de transferencia (ARNt), de los cuales hay al menos uno por cada uno de los 20 aminoácidos comunes. En un extremo, los ARNt presentan tres nucleótidos, el anticodón, que se aparea en forma específica con un grupo específico de codones. En el extremo opuesto al anticodón hay un sitio para la unión covalente de un aminoácido.
La producción de aminoacil-tRNA (ARNt con su aminoácido correspondiente) corre por cuenta de aminoacil tRNA sintetasas. Hay al menos 20 tipos, y cada tipo activa y une con su aminoácido específico a un tipo - o serie de tipos - de ARNt.
Los ribosomas son agregados de ARN y proteínas. Se pueden separar en dos subunidades, llamadas subunidad mayor y menor. Los ribosomas contienen sitios de unión al ARNm, al ARNt y a una serie de factores proteicos. En los ribosomas se cataliza la formación del enlace peptídico entre el polipéptido en formación y el aminoácido entrante.
Una serie de factores proteicos, usualmente unidos a los ribosomas, son reuqeridos para la correcta iniciación de la síntesis, para la unión de aminoacil tRNAs entrantes y para el movimiento del ribosoma respecto al ARNm tras cada nuevo agregado de aminoácido (translocación).
VI- CÓDIGO GENÉTICO:
El código genético establece la correspondencia entre una serie de nucleótidos del ARN mensajero a traducir y la secuencia de aminoácidos en la cadena polipeptídica resultante.
El ARN contiene los ribonucleótidos de Adenina, Citidina, Guanina y Uracilo; el ADN contiene desoxirribonuclótidos de Adenina, Citidina, Guanina y Timina. Cada nucleótido tomado por separado podría codificar en total 4 de los 20 aminoácidos posibles, de una proteína. Por lo tanto, se requiere un grupo de nucleótidos para representar cada aminoácido, y deberán formarse por lo menos 20 "palabras", representando cada una uno de los veinte aminoácidos.
Si se usaran dos nucleótidos para representar a cada aminoácido, se podrán codificar sólo 16 aminoácidos (es decir, 42), por lo que un mínimo de tres nucleótidos deben tomarse por vez para tener la capacidad de codificar todos los aminoácidos. Con tres nucleótidos podrán formarse 64 (43) combinaciones posibles de los cuatro nucleótidos existentes. El código genético actual es, en efecto, un código de tripletes, en el cual se leen los nucleótidos en grupos de a tres a partir de un punto de partida fijo.
De los 64 codones posibles del código, 61 especifican aminoácidos, quedando tres que tienen la función de marcar el fin de la traducción. De los aminoácidos con varios codones (codones denominados por ende sinónimos), hay diferentes agrupaciones: Por un lado, tres aminoácidos son codificados por 6 codones; en el otro extremo, dos aminoácidos (Met y Trp)tienen sólo un codón. A la existencia de redundancias se la llama degeneración del código.
La degeneración del código no es aleatoria. Como puede observarse en la tabla, los codones sinónimos se agrupan en familias que comparten la misma tercera posición: Por ejemplo, la valina es codificada por los codones CTT, CTC, CTA Y CTG, lo cual puede formularse como CTN (aNy). Cuando la familia de cuatro codones es compartida por dos aminoácidos, tampoco es una asignación aleatoria: La asparagina y la lisina son codificados por AAT y AAC, y AAA y AAG respectivamente, lo cual implica que la tercera posición de la lisina es una purina y la tercera posición de la asparagina es una pirimidina. De tal modo, una mutación en la tercera posición que implique una transición (de A a G o de T a C) no cambiará al aminoácido implicado. La síntesis de proteínas en todo tipo de células empieza con el aminoácido metionina, que cuando oficia de inicador es codificado en general con AUG (ATG en ADN). Los codones UAA, UGA, and UAG constituyen el terminador, que marca el extremo carboxiloterminal de la cadena proteica. A la secuencia de codones que va de un comienzo específico hasta un codón de terminación se le llama marco de lectura, en la cual no existe señal molecular que indique cuándo termina un codón y empieza el otro. Esta ausencia de "signos de puntuación" implica que la inserción o deleción de un nucleótido de la secuencia alterará seriamente el significado de toda la secuencia subsiguiente.
El significado de cada codón es el mismo en la mayoría de los organismos, lo cual constituye un argumento fuerte a favor de que todos descendemos del un mismo organismo. Ha de destacarse, no obstante, que si bien el código genético es universal, existen excepciones. Por ejemplo, en las mitocondrias humanas, ATA y ATG codifican ambas metionina, y TGA codifica triptofano. En general, las variaciones son pequeñas variaciones en torno a aminoácidos cercanos en el código (nótese que en el caso mencionado, lo que se hace es agregar un codón a la metionina y al triptofano). No son variaciones drásticas, que debieron haberse hecho imposibles ni bien empezara a funcionar el código actual.







No hay comentarios.:
Publicar un comentario